Die Plattform heißt MOSAIC (Multiscale Origami Structures as Interface for Cells). Origami heißt eigentlich "Papierfalten". Um Faltung geht es hier auch, aber um DNA-Faltung. Aus langen DNA-Fragmente, die sich selbstorganisiert nach einem genauen Plan in eine 100 Nanometer lange und 50 Nanometer breite Platten falten wurden Stecktafeln gestaltet, die an bestimmten Stellen Signalmoleküle binden können. Die Wissenschaftler haben Signalmoleküle im Abstand von 5 Nanometern auf kleinste Stecktafeln aufgebracht. Auf Zellträger (Microarrays) haben die Forscher passende DNA-Fundamente für die DNA-Stecktafeln aufgedruckt, an die sie nun binden konnten. So gelang es den Forschern, eine Vielzahl an Stecktafeln auf einem Microarray zu befestigen.
In einer Machbarkeitsstudie haben die Wissenschaftler die Funktionsfähigkeit ihrer Plattform getestet, indem sie Zellen einer gängigen Brustkrebs-Zelllinie (MCF-7) als Zellmodell auf ihre Reaktionsfähigkeit gegenüber Liganden (Signalmolekülen des Systems) getestet haben. Dafür wurden die Stecktafeln mit EGF-Molekülen (Liganden für den sogenannten Epithelial Growth Factor (EGFR)) konstruiert und hiernach MCF-7-Zellen aufgebracht. Die durch Bindung (Phosphorylierung) aktivierten Rezeptoren der einzelnen MCF-7-Zellen wurden durch Antikörperfärbung sichtbar gemacht. 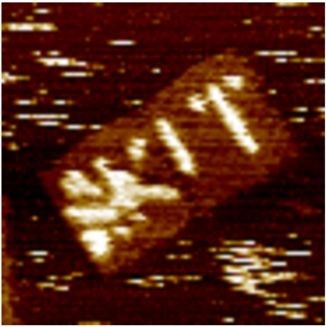
Mit hoher Genauigkeit lassen sich Moleküle mit dem MOSAIC-Verfahren auf einer Stecktafel von etwa 50 mal 100 Nanometer Größe positionieren. (Bild: KIT)
In dieser Machbarkeitsstudie konnte quantitativ gezeigt werden, dass auf unterschiedlich dicht besetzte Stecktafeln auf dem Zellträger auch die Zellen unterschiedlich intensiv reagierten. Auf den Steckplätzen können natürlich auch verschiedene Liganden aufgebracht werden. Im nächsten Schritt wollen die Forscher untersuchen, ob sie mit mehreren Liganden mehrere Rezeptoren der Zellen ansprechen können, ob dies in Kombination nacheinander geschieht und welchen Abstand die Liganden aufweisen müssen, um vom Rezeptor erfasst zu werden. Auch Zellreaktionen downstream lassen sich kombinieren, wenn z.B. Zellen gentechnisch mit einem Fluoreszenzprotein versehen werden, das z.B. immer dann produziert und sichtbar wird, wenn in der Zelle durch die Aktivierung eines/mehrerer Rezeptoren am Ende ein Transkriptionsfaktor aktiviert worden ist.
So könnten sich Krankheiten, die z.B. auf eine Fehlfunktion von Zellrezeptoren oder der Signalwege zurückzuführen sind, untersuchen lassen.
Originalpublikation:
Alessandro Angelin, Simone Weigel, Ruben Garrecht, Rebecca Meyer, Jens Bauer, Ravi Kapoor Kumar, Michael Hirtz & Christof M. Niemeyer (2015):
Multiscale Origami Structures as Interface for Cells. Angew. Chem. Int. Ed. 54, 15813–15817. DOI: 10.1002/anie.201509772
Quellen:
http://www.kit.edu/kit/pi_2016_038_der-grammatik-biologischer-zellen-auf-der-spur.php
http://www.ibg.kit.edu/ibg1/
http://www.ibg.kit.edu/ibg1/212.php
Diese Seite drucken
Donnerstag, 17 März 2016 16:03
MOSAIC: Neue Plattform zur Analyse von Signaltransduktionen in Zellen Empfehlung
Wissenschaftler vom Institut für biologische Grenzflächen des Karlsruher Institut für Technologie (KIT) unter der Leitung von Prof. Christoph Niemeyer haben eine Plattform entwickelt, mit der sich grundlegende Aspekte der frühen Stadien der Signaltransduktion in lebenden Zellen untersuchen lassen.
